Conceito da linguagem e manipulação de objetos
O software R
Como já discutido na aula motivacional para o curso, R é uma linguagem e ambiente para computação estatística e gráfica, disponível sob os termos da licença GNU de software livre. Criada em 1996 por Robert Gentleman and Ross Ihaka, o R pode ser considerado um diferente implementação de S, a linguagem comercial desenvolvida pela Bell Laboratories, que antecede o R.
Pode-se destacar diversos tópicos que fazem do R a excelência em software para manipulação de dados, análise estatística e visualização gráfica, porém listamos apenas alguns abaixo:
- Eficaz em manipulação e armazenamento de dados;
- Grande conjunto de recursos para calculos vetoriais e matriciais;
- Diversas ferramentas para análise de dados;
- Ampla suíte de funções para visualização de dados, que podem ser exportados para diversos formatos;
- Linguagem de programação simples e eficaz (loops, condicionais, funções recursivas, etc.); e
- Simples construção e disponibilização de pacotes com funções específicas, via CRAN (Comprehensive R Archive Network)
Explore o sítio do projeto R e o espelho c3sl UFPR da rede global de arquivos R.
- Projeto R (https://www.r-project.org/);
- Repositório R (http://cran-r.c3sl.ufpr.br/)
Outros sites interessantes sobre iniciativas com o R são:
- ROpenSci - R Open Science (https://ropensci.org/);
- RConsortium - grupo para prestar suporte a Comunidade R (https://www.r-consortium.org/);
- MRAN - Microsoft R Application Network (https://mran.revolutionanalytics.com/)
- FOAS - Foundation for Open Access Statistics (http://www.foastat.org/)
RStudio IDE
O ambiente de desenvilvimento integrado - IDE (Intergrated DEvelopment Environment) RStudio é um produto da empresa RStudio (https://www.rstudio.com/) que, como o próprio nome diz, é um ambiente de desenvolvimento integrado de códigos R. O RStudio IDE permite o desenvolvimento de códigos de outras linguagens também, porém diversas funcionalidade são otimizadas para R.
O download do RStudio IDE pode ser feito pelo endereço https://www.rstudio.com/products/rstudio/download/ com versões disponíveis para diversas plataformas (Linux, Windows e MAC).
No curso utilizaremos essencialmente o RStudio por facilidade, mas existem outros editores e o usuário de R tem a liberdade para escolher o que mais lhe agrada. Para a escolha de um editor destacamos algumas ferramentas que são indispensáveis para um bom desenvolvimento de programação R:
- Destaque de código;
- Destaque de marcadores de ambiente (parênteses, chaves e colchetes);
- Identação automática;
- Auto-completar de função e objetos; e
- Interação para edição de documentos híbridos (R+Markdown e R+LaTeX).
Elementos da interface
Ao instalar o RStudio, temos a interface do programa com a seguinte estrutura descrita abaixo:
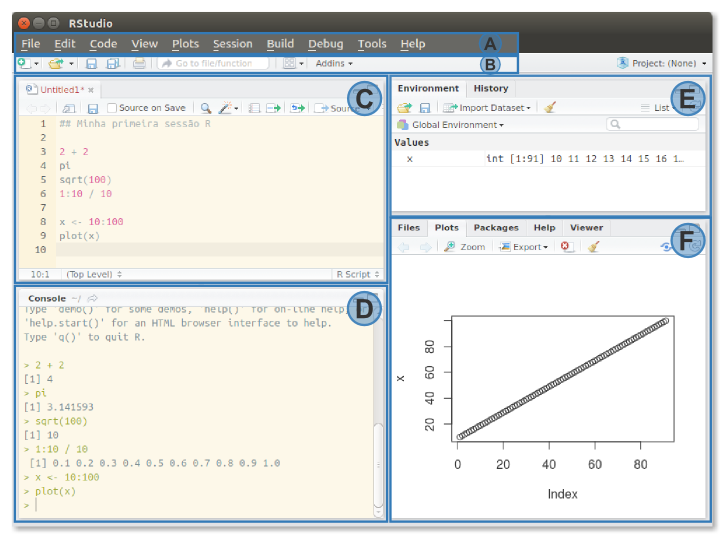
em que:
- (A): Barra de navegação
- Contém todas as funcionalidades do RStudio IDE desde criar, abrir arquivos e ferramentas para edição de códigos (desfazer, refazer, etc.) além de inserir seções até as configurações globais e documentos de ajuda sobre o programa.
- (B): Barra de acesso rápido
- Esses são apenas atalhos rápidos para algumas rotinas mais utilizadas pelos usuários como criar, abrir, salvar arquivos, por exemplo.
- (C): Edição de scripts
- Este é o ambiente no qual escrevemos nossos códigos. Todo o código de uma análise é escrito nesse campo. Como já dito o RStudio não é um editor somente de R, arquivos R e de outras linguagens também podem ser editados aqui. Note que vários arquivos podem ser abertos simultaneamente, eles ficarão dispostos em abas nesse painel.
- (D): Console R
- Nesse campo temos o R propriamente dito, o chamamos de console R. Tudo que for escrito aqui será automaticamente interpretado. O que fazemos é enviar os códigos escritos em (C) para cá, com o atalho de teclado
Crtl+R, ou atalho da interface. - (E): Visualização de objetos e códigos elaborados
- Esse painel é composto por duas abas. A primeira, Environment, exibe as variáveis de ambiente criadas durante a sessão e a segunda, History exibe toda a sequência de códigos interpretada pelo console na sessão.
- (F): Visualização gráficos, arquivos, pacotes e ajuda
- Neste painel têm-se diversas abas listadas abaixo: * Files: explorador de arquivos do R; * Plots: visualização de gráficos interpretados pelo console (D); * Packages: lista de pacotes instalados e atalho para instalação de pacotes; * Help: lista de ajuda para funções do R e atalho para suportes online do RStudio; e * Viewer: Visualizador de outputs web, como páginas html, aplicativos Shiny, apresentações Rpres ou documentos pdf.
Aqui foram listados, brevemente, alguns aspectos da interface, todavia seja curioso e explore as demais funcionalidades do RStudio IDE.
Alguns atalhos
Toda a edição de scripts realizada no RStudio IDE pode ser otimizada utilizando atalhos do teclado para algumas rotinas. Quanto menos você precisar do mouse mais rápida e eficientemente você programa.
Todos os atalhos do RStudio podem ser visualizados no menu de navegação (A)->Tools->Keyboard Shortcuts Help, ou ainda pelo atalho Alt+Shift+K. Os atalhos indispensáveis são:
Alt+-: Sinal de atribuir (<-);Ctrl+r: Executar linha/região do script;Ctrl+i: Indentar região; eCtrl+Shift+C: Comentar linha/região.
Aritmética e objetos
Operações aritméticas
O R pode ser usado como calculadora, todas as operações matemáticas básicas estão disponíveis em R.
2 + 2 # Soma## [1] 44 - 2 # Subtração## [1] 212 * 42 # Multiplicação## [1] 50454 / 30 # Divisão## [1] 1.85^3 # Potência## [1] 1255**3 # Potência## [1] 125E claro, expressões numéricas também podem ser feitas. Respeita-se a hierárquia matemática primeiro potência < multiplicação < soma, porém essa ordenação pode ser contralada por parênteses.
2 + 2 * 4 + 4/2 # Hierarquia matemática## [1] 122 + 2 * (4 + 4)/2 # Primeiro a soma dos 4's## [1] 10(2 + 2) * 4 + 4/2 # Primeiro a soma dos 2's## [1] 18(2 + 2) * (4 + 4)/2 # Primeiro as somas## [1] 16((2 + 2) * (4 + 4)/2)/2 # Vários parenteses## [1] 8Operações matematicamente incorretas ou não exatas também tem seu valor representado em R.
0/0 # Impossível, não existe (Not a Number - NaN)## [1] NaN1/0 # Indefinido, positivo de infinito (Inf)## [1] Inf-1/0 # Indefinido, negativo de infinito (-Inf)## [1] -InfNA # Valor não disponível (Not Available - NA)## [1] NAOperações lógicas
As operações descritas acima são operações matemáticas simples, porém em muitas situações deseja-se realizar testes lógicos, e.g. o peso do paciente é maior de 60Kg, abaixo exemplificamos como realizar esses testes.
2 == 4 # Igualdade## [1] FALSE2 > 4 # Desigualdade (maior)## [1] FALSE2 < 4 # Desigualdade (menor)## [1] TRUE2 >= 4 # Desigualdade (maior igual)## [1] FALSE2 <= 4 # Desigualdade (menor igual)## [1] TRUEE claro expressões lógicas também podem ser realizadas.
(2 + 2) >= 4## [1] TRUE(2 + 2)/2 < 4## [1] TRUEOutro operador lógico muito útil é o de negação !, esse operador inverte um resultado lógico
!2 == 4## [1] TRUE!2 > 4## [1] TRUE!2 < 4## [1] FALSECriando objetos
O R é uma linguagem orientada a objetos, ou seja, objetos (valores, vetores, matrizes, listas, funções, etc.) são armazenadas em espaços de memória de seu computador, para posteriormente serem utilizados. Atribui-se algo a um objeto pelo operador <-.
x <- 2 # Atribuindo o valor 2 ao objeto x
y <- 4 # Atribuindo o valor 2 ao objeto y
x + y## [1] 6z <- x * y # Atribuindo ao z o valor da multiplicação de x por y
z## [1] 8a <- 2 == 2 # Valores lógicos também podem ser armazenados em objetos
a## [1] TRUEIsso mesmo, em R a atribuição é realizada de forma muito simples!
Tipos de objetos
O R tem uma gama muito grande de classes de objetos. Todo objeto possui uma classe que o define. Usuários de R devem conhecer bem as classes de seus objetos para coordenar funções apropriadas a eles.
Use e abuse das funções class(), que exibe a classe do objeto e str(), que mostra sua estrutura.
# Verificando a estrutura dos objetos criados
str(x)## num 2class(x)## [1] "numeric"str(a)## logi TRUEclass(a)## [1] "logical"# Classes comuns no R
"string"; class("string") # character## [1] "string"## [1] "character"100L; class(100L) # integer## [1] 100## [1] "integer"100; class(100) # numeric## [1] 100## [1] "numeric"TRUE; class(TRUE) # logical## [1] TRUE## [1] "logical"Vetores
O R é uma linguagem vetorial, ou seja, operações com objetos dessa classe são realizadas diretamento, sem a necessidade de loops (for, por exemplo).
A criação de vetores é feita, em geral, com a função c() de combinar valores em vetores ou listas.
x <- c(2, 4, 6, 8, 10, 12)
x## [1] 2 4 6 8 10 12# O que é x?
str(x)## num [1:6] 2 4 6 8 10 12class(x)## [1] "numeric"Vetores também podem ser criados com as funções rep() e seq(), que fazem repetições e sequências respectivamente e com o operador : que faz a sequência de incremento 1 entre dois valores.
y <- 1:6
y## [1] 1 2 3 4 5 6E claro, as operações com os vetores.
x * 2## [1] 4 8 12 16 20 24x / y## [1] 2 2 2 2 2 2As operações lógicas seguem da mesma forma, todavia podemos introduzir um novo operador o %in%, esse é o operador “pertence”.
x == y # x é igual a y, elemento a elemento?## [1] FALSE FALSE FALSE FALSE FALSE FALSE2 %in% y # 2 pertence ao conjunto de y?## [1] TRUEx %in% y # x pertence ao conjunto y, elemento a elemento?## [1] TRUE TRUE TRUE FALSE FALSE FALSEE a seleção de valores do vetor é feita com os operadores [].
y[1] # Seleciona o 1º elemento## [1] 1y[c(1, 3)] # Seleciona o 1º e quinto elementos## [1] 1 3y[y > 3] # Seleciona apenas os y que são maiores que 3## [1] 4 5 6y[x %in% y] # Seleciona apenas os y que satizfazem x %in% y## [1] 1 2 3Matrizes
Matrizes representam um conjunto de valores organizados em linhas e colunas. Matrizes são criadas com a função matrix(), onde informamos os valores que faram parte da matriz e a dimensão dessa matriz, os argumentos da função são (não se preocupe com todos os argumentos, alguns tem um valor padrão! Discutiremos funções adiante).
# Argumentos da função matrix
# help(matrix)
args("matrix")## function (data = NA, nrow = 1, ncol = 1, byrow = FALSE, dimnames = NULL)
## NULLAgora criando matrizes
# Matrizes com 10 elementos de dimensão 2x5
matrix(data = 1:10, nrow = 2, ncol = 5)## [,1] [,2] [,3] [,4] [,5]
## [1,] 1 3 5 7 9
## [2,] 2 4 6 8 10matrix(data = 1:10, nrow = 2, ncol = 5, byrow = TRUE)## [,1] [,2] [,3] [,4] [,5]
## [1,] 1 2 3 4 5
## [2,] 6 7 8 9 10# Matrizes com 18 elementos de dimensão 3x6
matrix(data = 1:18, nrow = 3)## [,1] [,2] [,3] [,4] [,5] [,6]
## [1,] 1 4 7 10 13 16
## [2,] 2 5 8 11 14 17
## [3,] 3 6 9 12 15 18matrix(data = 1:18, nrow = 3, byrow = TRUE)## [,1] [,2] [,3] [,4] [,5] [,6]
## [1,] 1 2 3 4 5 6
## [2,] 7 8 9 10 11 12
## [3,] 13 14 15 16 17 18A seleção de elementos da matriz também é feita com o operador [], porém agora temos duas dimensões para seleção, as linhas e as colunas.
# Matrizes com 18 elementos de dimensão 3x6
ma <- matrix(data = 1:18, nrow = 3, byrow = TRUE)
ma## [,1] [,2] [,3] [,4] [,5] [,6]
## [1,] 1 2 3 4 5 6
## [2,] 7 8 9 10 11 12
## [3,] 13 14 15 16 17 18# Estrutura do objeto
str(ma)## int [1:3, 1:6] 1 7 13 2 8 14 3 9 15 4 ...ma[1, 3] # Elemento da linha 1 e coluna 3## [1] 3ma[, 3] # Elementos da coluna 3 (é um vetor)## [1] 3 9 15ma[1, ] # Elementos da linha 1 (é um vetor)## [1] 1 2 3 4 5 6O foco deste curso não será na computação. Portanto não apresentaremos as operações com matrizes mas saiba que existem, adição e multiplicação de matrizes, transposta, inversa, produto interno, traço, entre outras operações podem facilmente realizadas com funções nativas do R.
Data frames
data.frames são a classe de objetos que mais utilizamos. Eles são a representação dos nossos dados em R. Podemos criar data.frames com a função data.frame. Assim como nas matrizes, veja seus argumentos e consulte o help da função.
# Argumentos da função data.frame
# help(data.frame)
args("data.frame")## function (..., row.names = NULL, check.rows = FALSE, check.names = TRUE,
## fix.empty.names = TRUE, stringsAsFactors = default.stringsAsFactors())
## NULL# Um exemplo de data.frame
data.frame(x = 1:6, y = 11:16, z = rep(0:1, 3))## x y z
## 1 1 11 0
## 2 2 12 1
## 3 3 13 0
## 4 4 14 1
## 5 5 15 0
## 6 6 16 1# Construindo data.frames com objetos criados
data.frame(x = x, y = y, z = x * y, w = x %in% y)## x y z w
## 1 2 1 2 TRUE
## 2 4 2 8 TRUE
## 3 6 3 18 TRUE
## 4 8 4 32 FALSE
## 5 10 5 50 FALSE
## 6 12 6 72 FALSEAgora que já sabemos como criar data.frames, criaremos um data.frame fictício para exemplificar a seleção de elementos de um objeto dessa classe, que pode realizada com os operadores $ ou ainda [].
# Criando dados fictícios de pacientes
da <- data.frame(
nome = c("João", "Maria", "José", "Pedro", "Ana", "Carla", "Paulo"),
idade = c(30, 22, 21, 40, 32, 18, 24),
sexo = c("M", "F", "M", "M", "F", "F", "M"),
imc = c(30.98, 37, 42.29, 27.59, 21.18, 25.23, 8.81),
hdl = c(68.89, 70.25, 15.59, 49.24, 50.79, 45.22, 56.65)
)
da## nome idade sexo imc hdl
## 1 João 30 M 30.98 68.89
## 2 Maria 22 F 37.00 70.25
## 3 José 21 M 42.29 15.59
## 4 Pedro 40 M 27.59 49.24
## 5 Ana 32 F 21.18 50.79
## 6 Carla 18 F 25.23 45.22
## 7 Paulo 24 M 8.81 56.65# Estrutura dos dados
str(da)## 'data.frame': 7 obs. of 5 variables:
## $ nome : Factor w/ 7 levels "Ana","Carla",..: 3 5 4 7 1 2 6
## $ idade: num 30 22 21 40 32 18 24
## $ sexo : Factor w/ 2 levels "F","M": 2 1 2 2 1 1 2
## $ imc : num 31 37 42.3 27.6 21.2 ...
## $ hdl : num 68.9 70.2 15.6 49.2 50.8 ...da[2, ] # Selecionando os dados da Maria## nome idade sexo imc hdl
## 2 Maria 22 F 37 70.25da[, 2] # Selecionando as idades## [1] 30 22 21 40 32 18 24da[, "idade"] # Selecionando as idades## [1] 30 22 21 40 32 18 24da$idade # Selecionando as idades## [1] 30 22 21 40 32 18 24da[, c("imc", "hdl")] # Selecionando as variáveis imc e hdl## imc hdl
## 1 30.98 68.89
## 2 37.00 70.25
## 3 42.29 15.59
## 4 27.59 49.24
## 5 21.18 50.79
## 6 25.23 45.22
## 7 8.81 56.65da[2, c("imc", "hdl")] # Selecionando as variáveis imc e hdl da Maria## imc hdl
## 2 37 70.25Note que a seleção de elementos é bastante similar a seleção no caso das matrizes. A principal diferença entre matrizes e data.frames no R é que data.frames permitem que cada coluna possua um vetores com elementos de classes diferentes (fatores, numéricos, inteiros, caracteres, lógicos, etc.), isso não é possível em matrizes.
Listas
As listas são os objetos mais flexíveis em R, elas permitem armazenar diversos formatos em um único objeto. Listas são criadas com a função list().
# Lista de vetores
list(a = 1:6, b = c("Olá", "mundo"), c = c(TRUE, FALSE, FALSE))## $a
## [1] 1 2 3 4 5 6
##
## $b
## [1] "Olá" "mundo"
##
## $c
## [1] TRUE FALSE FALSE# Lista com objetos de diferentes tipos
la <- list(ma = ma, da = da, x = x, y = y)
la## $ma
## [,1] [,2] [,3] [,4] [,5] [,6]
## [1,] 1 2 3 4 5 6
## [2,] 7 8 9 10 11 12
## [3,] 13 14 15 16 17 18
##
## $da
## nome idade sexo imc hdl
## 1 João 30 M 30.98 68.89
## 2 Maria 22 F 37.00 70.25
## 3 José 21 M 42.29 15.59
## 4 Pedro 40 M 27.59 49.24
## 5 Ana 32 F 21.18 50.79
## 6 Carla 18 F 25.23 45.22
## 7 Paulo 24 M 8.81 56.65
##
## $x
## [1] 2 4 6 8 10 12
##
## $y
## [1] 1 2 3 4 5 6# Estrutura da lista
str(la)## List of 4
## $ ma: int [1:3, 1:6] 1 7 13 2 8 14 3 9 15 4 ...
## $ da:'data.frame': 7 obs. of 5 variables:
## ..$ nome : Factor w/ 7 levels "Ana","Carla",..: 3 5 4 7 1 2 6
## ..$ idade: num [1:7] 30 22 21 40 32 18 24
## ..$ sexo : Factor w/ 2 levels "F","M": 2 1 2 2 1 1 2
## ..$ imc : num [1:7] 31 37 42.3 27.6 21.2 ...
## ..$ hdl : num [1:7] 68.9 70.2 15.6 49.2 50.8 ...
## $ x : num [1:6] 2 4 6 8 10 12
## $ y : int [1:6] 1 2 3 4 5 6Note que la é uma lista com 4 elementos, para selecionarmos elementos dessa lista usamos o operador [[]].
la[[1]] # Primeiro elemento da lista## [,1] [,2] [,3] [,4] [,5] [,6]
## [1,] 1 2 3 4 5 6
## [2,] 7 8 9 10 11 12
## [3,] 13 14 15 16 17 18la[["da"]] # Elemento 'da' da lista## nome idade sexo imc hdl
## 1 João 30 M 30.98 68.89
## 2 Maria 22 F 37.00 70.25
## 3 José 21 M 42.29 15.59
## 4 Pedro 40 M 27.59 49.24
## 5 Ana 32 F 21.18 50.79
## 6 Carla 18 F 25.23 45.22
## 7 Paulo 24 M 8.81 56.65la[c("x", "y")] # Sub-lista com x e y## $x
## [1] 2 4 6 8 10 12
##
## $y
## [1] 1 2 3 4 5 6# Agora para selecionar as idades do elemento 'da' da lista 'la'
la[["da"]][, "idade"]## [1] 30 22 21 40 32 18 24# E a segunda linha do elemento 'ma' da lista 'la'
la[["ma"]][2, ]## [1] 7 8 9 10 11 12Funções
Uma das grandes vantagens em linguagens de programação é que podemos criar nossas próprias funções e com isso controladas toda a rotina de análise. Em R não pe diferente, podemos criar nossas próprias função, bem como utilizar as que já existem, como fizemos até agora.
Funções em R são construídas com a estrutura de programação function. Abaixo exemplificamos como se cria uma função em R.
# Função que soma 2 ao elemento
fx <- function(a) {
(a + 2)
}
# O que é fx?
class(fx)## [1] "function"# Usando a minha função
fx(2)## [1] 4fx(5)## [1] 7# fx("a") Erro
# Função com mais de um argumento
fx2 <- function(a, b) {
a + b
}
fx2(5, 4)## [1] 9fx2(3, 2)## [1] 5# fx2(3) Erro
# Função com argumento padrão (default)
fx2 <- function(a, b = 2) {
a + b
}
fx2(5)## [1] 7fx2(6)## [1] 8fx2(6, 5)## [1] 11E é dessa forma que são construídas as funções em R. Eventualmente precisamos escrever nossas função, porém para análises simples todas as funções já existem no R. Para utilizá-las utilize a sintaxe funcao(argumentos) e não dispense ler sua documentação help("funcao").
Abaixo mostramos algumas funções para cálculo de estatísticas básicas para o conjunto de dados fictício da.
mean(da$hdl) # média de hdl## [1] 50.94714median(da$hdl) # mediana de hdl## [1] 50.79var(da$hdl) # variância## [1] 335.4893sd(da$hdl) # desvio padrão## [1] 18.31637min(da$hdl) # valor mínimo## [1] 15.59max(da$hdl) # valor máximo## [1] 70.25quantile(da$hdl) # Quantis## 0% 25% 50% 75% 100%
## 15.59 47.23 50.79 62.77 70.25Pacotes em R
O R possui uma comunidade muito ativa de desenvolvedores e com sua facilidade de programação diversos pacotes de função são disponibilizados aos usuários diariamente. O principal repositório de disponibilização de pacotes é o CRAN e é por lá que fazemos a instalação de pacotes adicionais.
Não se preocupe em ir pro site, baixar arquivo e depois instalar. A comunidade R já resolveu isso, para instalação de pacotes do CRAN basta utilizar a função install.packages().
# help(install.packages)
# Instalando o pacote rmarkdown, que utilizaremos ao final do curso
install.packages("rmarkdown")Entrada de dados
Como vimos na seção Data Frames, nosso dados são representados em R como data.frames e podemos digitar cada dado de nossa eventual planilha para o R, como fizemos com o exemplo da. Todavia isso acaba se tornando inviável rapidamente, e surge a necessidade de entrar com dados no R via arquivos externos, é disso que tratamos na próxima seção.
De arquivos externos
O local mais comum para armazenamento de conjuntos de dados não tão grandes, é em planilhas eletrônicas (LibreOffice Calc, Excel, etc.), esses arquivos tem extensão xlsx, xls, odt entre outras, esses arquivos são arquivos binários, que são interpretador por um programa e exibem uma interface para edição. A leitura de arquivos binários em R é possível, porém recomendamos que para leitura o usuário exporte os dados de sua planilha para um formato de texto pleno (.csv, .tsv, txt).
Para leitura de dados em texto pleno utiliza-se a função read.table.
# head(read.table)
args(read.table)## function (file, header = FALSE, sep = "", quote = "\"'", dec = ".",
## numerals = c("allow.loss", "warn.loss", "no.loss"), row.names,
## col.names, as.is = !stringsAsFactors, na.strings = "NA",
## colClasses = NA, nrows = -1, skip = 0, check.names = TRUE,
## fill = !blank.lines.skip, strip.white = FALSE, blank.lines.skip = TRUE,
## comment.char = "#", allowEscapes = FALSE, flush = FALSE,
## stringsAsFactors = default.stringsAsFactors(), fileEncoding = "",
## encoding = "unknown", text, skipNul = FALSE)
## NULLAntes de utilizar essa função verifique como o arquivo está disposto, faça isso visualizando seu arquivo em um editor de texto, pode ser até no RStudio. Feito isso identifique
- Existe um cabeçalho com os nomes das colunas?
Se sim, coloqueheader = TRUE; - Qual o caracter que está separando os dados?
- se é uma tabulação (espaço entre valores) coloque
sep = "\t"; - se é uma ponto e vírgula (;) coloque
sep = ";"; - se é uma vírgula (;) coloque
sep = ","; - se outro caracter coloque
sep = "outro caracter" - Qual o separador decimal utilizado?
- se é ponto, padrão americano coloque
dec = "." - se é vírgula, padrão brasileiro coloque
dec = "," - Existe nomes que contém aspas?
- se sim, coloque
quote = ""
Estes são os principais argumentos da função, caso a leitura for incorreta procure identiicar o erro visualizando o arquivo e investigando os demais argumentos. Faremos o exercício de leitura de dados com o exemplo real de dados de médicos residentes por município, os dados estão disponíveis para download em medicos.csv.
# Lendo os dados sobre médicos residentes
# Baixado do sítio <http://www.ipeadata.gov.br/>
# Baixe em <stats-cwr.github.io/stats4med/data/medicos.csv>
# Primeiro veja como o arquivo está disposto (abra o csv em um bloco de
# notas ou no RStudio). Aqui veremos sua estutura com a função readLines
head(readLines("./data/medicos.csv"), 10)## [1] "Médicos residentes (por mil habitantes)"
## [2] "Sigla;Código;Município;1991;2000;"
## [3] "AC;1200013;Acrelândia;0;0;"
## [4] "AC;1200054;Assis Brasil;0;0;"
## [5] "AC;1200104;Brasiléia;0;0,506;"
## [6] "AC;1200138;Bujari;0;0;"
## [7] "AC;1200179;Capixaba;0;0;"
## [8] "AC;1200203;Cruzeiro do Sul;0,156;0,528;"
## [9] "AC;1200252;Epitaciolândia;0;0;"
## [10] "AC;1200302;Feijó;0;0;"Note que a primeira linha tem apenas um título para o arquivo e não pertence ao conjunto de dados, portanto devemos pular a 1º linha. Na segunda linha temos um cabeçalho que representa as caracteriticas que foram observadas em cada município. Os dados estão separados por ponto e vírgula ;. O separador decimal é do padrão brasileiro com vírgula ,. E temos nomes de cidades que utilizam apóstofro '. Com base nessas informações a leitura dos dados será
# Lendo os dados
dados <- read.table(
"./data/medicos.csv", # o caminho para o .csv
header = TRUE, # tem cabeçalho
sep = ";", # Separador ;
dec = ",", # Decimais com ,
quote = "", # Nada define caracteres no .csv
skip = 1, # Pula a primeira linha
encoding = "UTF-8" # Codificação de caracteres uft-8
)
# Verificando se a leitura foi correta
str(dados)## 'data.frame': 5596 obs. of 6 variables:
## $ Sigla : Factor w/ 27 levels "AC","AL","AM",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ Código : int 1200013 1200054 1200104 1200138 1200179 1200203 1200252 1200302 1200328 1200336 ...
## $ Município: Factor w/ 5315 levels "Abadia de Goiás",..: 27 396 709 751 1022 1414 1603 1678 2499 2769 ...
## $ X1991 : num 0 0 0 0 0 0.156 0 0 0 0 ...
## $ X2000 : num 0 0 0.506 0 0 0.528 0 0 0 0 ...
## $ X : logi NA NA NA NA NA NA ...# Podemos excluir a última coluna, pois essa só foi adicionada porque as
# linhas terminam em ;
dados <- dados[, -6]
# Podemos renomear as colunas para facilitar a manipulação
colnames(dados) <- c("estado", "codigo", "municipio", "med1", "med2")
# Verificando novamente a estrutura
str(dados)## 'data.frame': 5596 obs. of 5 variables:
## $ estado : Factor w/ 27 levels "AC","AL","AM",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ codigo : int 1200013 1200054 1200104 1200138 1200179 1200203 1200252 1200302 1200328 1200336 ...
## $ municipio: Factor w/ 5315 levels "Abadia de Goiás",..: 27 396 709 751 1022 1414 1603 1678 2499 2769 ...
## $ med1 : num 0 0 0 0 0 0.156 0 0 0 0 ...
## $ med2 : num 0 0 0.506 0 0 0.528 0 0 0 0 ...E assim temos o conjunto de dados medicos.csv devidamente disponível no R.
Para dados de texto pleno a principal função é a read.table, todavia podemos procurar por outras funções de leitura.
# Funções que contém 'read' no nome
apropos("read")## [1] ".readRDS" "read_chunk" "read_demo"
## [4] "read_rforge" "read.csv" "read.csv2"
## [7] "read.dcf" "read.delim" "read.delim2"
## [10] "read.DIF" "read.fortran" "read.ftable"
## [13] "read.fwf" "read.socket" "read.table"
## [16] "readBin" "readChar" "readCitationFile"
## [19] "readline" "readLines" "readRDS"
## [22] "readRenviron" "Sys.readlink"Extraindo estatísticas simples
Agora que já sabemos ler corretamente um conjunto de dados, selecionar diversos objetos e coordenar funções no R, podemos juntar todo esse conhecimento para extrair estatísticas simples da base de dados que acabamos de ler.
# Resumo dos dados
summary(dados)## estado codigo municipio med1
## MG : 853 Min. :1100015 Bom Jesus : 5 Min. :0.0000
## SP : 646 1st Qu.:2512175 Bonito : 4 1st Qu.:0.0000
## RS : 499 Median :3146280 Planalto : 4 Median :0.0000
## BA : 421 Mean :3253647 Santa Helena: 4 Mean :0.2317
## PR : 403 3rd Qu.:4119264 Santa Inês : 4 3rd Qu.:0.2845
## SC : 296 Max. :5300108 Santa Luzia : 4 Max. :6.8710
## (Other):2478 (Other) :5571 NA's :89
## med2
## Min. :0.0000
## 1st Qu.:0.0000
## Median :0.0000
## Mean :0.2708
## 3rd Qu.:0.4210
## Max. :7.2730
## NA's :89Familía apply
Em R existe uma família de função que usufruem da vantagem do R ser uma linguagem vetorial, as funções apply. Essas funções aplicam alguma função a um conjunto predefinido em algum objeto, e.g. calcular a média de idade das mulheres no conjunto de dados fictícios da.
São diversas as funções da família apply, abaixo temos uma lista mais que completa.
# Funções que terminam em apply
apropos("apply$")## [1] ".mapply" "apply" "dendrapply" "eapply"
## [5] "grid.DLapply" "kernapply" "lapply" "mapply"
## [9] "rapply" "sapply" "tapply" "vapply"Aqui utilizaremos somente as funções:
apply(): opera em matrizes e data.frames.lapply(): opera em vetores e listas (data.frame também é uma lista).sapply(): idem àlapplymas simplifica quando possível.tapply(): um vetor reposta em função de (combinação de) estratos.
Para exemplificar a utilização dessas funções voltemos ao conjunto de dados dados. Lembre-se que esse conjunto de dados contém NA's, portanto deve-se retirá-los, faremos isso com argumentos das funções (o na.rm), mas pode-se retirar da base com na.omit ou na.exclude.
# Escolhe as variáveis de interesse
vars <- c("med1", "med2")
# Médias de idade, imc e hdl
apply(X = dados[, vars], MARGIN = 2, FUN = mean, na.rm = TRUE)## med1 med2
## 0.2316806 0.2707808# Mínino, 1 quartil, mediana, média, 3º quartil e máximo
apply(X = dados[, vars], MARGIN = 2, FUN = summary)## med1 med2
## Min. 0.0000 0.0000
## 1st Qu. 0.0000 0.0000
## Median 0.0000 0.0000
## Mean 0.2317 0.2708
## 3rd Qu. 0.2845 0.4210
## Max. 6.8710 7.2730
## NA's 89.0000 89.0000# Mínino, 1 quartil, mediana, média, 3º quartil e máximo
lapply(X = dados[, vars], FUN = summary)## $med1
## Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
## 0.0000 0.0000 0.0000 0.2317 0.2845 6.8710 89
##
## $med2
## Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
## 0.0000 0.0000 0.0000 0.2708 0.4210 7.2730 89# Mínino, 1 quartil, mediana, média, 3º quartil e máximo
sapply(X = dados[, vars], FUN = summary)## med1 med2
## Min. 0.0000 0.0000
## 1st Qu. 0.0000 0.0000
## Median 0.0000 0.0000
## Mean 0.2317 0.2708
## 3rd Qu. 0.2845 0.4210
## Max. 6.8710 7.2730
## NA's 89.0000 89.0000#-------------------------------------------
# Agora médias estratificadas por sexo
tapply(X = dados[, "med1"], INDEX = dados[, "estado"],
FUN = mean, na.rm = TRUE)## AC AL AM AP BA CE
## 0.02281818 0.11896040 0.08443548 0.05056250 0.11662651 0.07042935
## DF ES GO MA MG MS
## 2.11400000 0.37345455 0.25871901 0.07854839 0.25471161 0.40357143
## MT PA PB PE PI PR
## 0.25975397 0.10627972 0.09723767 0.10690811 0.06149321 0.25226817
## RJ RN RO RR RS SC
## 0.82318681 0.08851807 0.15250000 0.45266667 0.32635760 0.28199317
## SE SP TO
## 0.05116000 0.41297519 0.17179137tapply(X = dados[, "med2"], INDEX = dados[, "estado"],
FUN = mean, na.rm = TRUE)## AC AL AM AP BA CE
## 0.17754545 0.15764356 0.14282258 0.08300000 0.13450120 0.12117935
## DF ES GO MA MG MS
## 2.04500000 0.42779221 0.23546281 0.07273733 0.30981477 0.23202597
## MT PA PB PE PI PR
## 0.28926190 0.12518881 0.09879821 0.12165946 0.05747059 0.31465163
## RJ RN RO RR RS SC
## 0.84309890 0.12803012 0.21711538 0.16513333 0.40568308 0.33447440
## SE SP TO
## 0.13041333 0.48426977 0.24969784Para treinar
Bases disponíveis
# Lista de conjuntos de dados para exercício
ls("package:datasets")## [1] "ability.cov" "airmiles"
## [3] "AirPassengers" "airquality"
## [5] "anscombe" "attenu"
## [7] "attitude" "austres"
## [9] "beaver1" "beaver2"
## [11] "BJsales" "BJsales.lead"
## [13] "BOD" "cars"
## [15] "ChickWeight" "chickwts"
## [17] "co2" "CO2"
## [19] "crimtab" "discoveries"
## [21] "DNase" "esoph"
## [23] "euro" "euro.cross"
## [25] "eurodist" "EuStockMarkets"
## [27] "faithful" "fdeaths"
## [29] "Formaldehyde" "freeny"
## [31] "freeny.x" "freeny.y"
## [33] "HairEyeColor" "Harman23.cor"
## [35] "Harman74.cor" "Indometh"
## [37] "infert" "InsectSprays"
## [39] "iris" "iris3"
## [41] "islands" "JohnsonJohnson"
## [43] "LakeHuron" "ldeaths"
## [45] "lh" "LifeCycleSavings"
## [47] "Loblolly" "longley"
## [49] "lynx" "mdeaths"
## [51] "morley" "mtcars"
## [53] "nhtemp" "Nile"
## [55] "nottem" "npk"
## [57] "occupationalStatus" "Orange"
## [59] "OrchardSprays" "PlantGrowth"
## [61] "precip" "presidents"
## [63] "pressure" "Puromycin"
## [65] "quakes" "randu"
## [67] "rivers" "rock"
## [69] "Seatbelts" "sleep"
## [71] "stack.loss" "stack.x"
## [73] "stackloss" "state.abb"
## [75] "state.area" "state.center"
## [77] "state.division" "state.name"
## [79] "state.region" "state.x77"
## [81] "sunspot.month" "sunspot.year"
## [83] "sunspots" "swiss"
## [85] "Theoph" "Titanic"
## [87] "ToothGrowth" "treering"
## [89] "trees" "UCBAdmissions"
## [91] "UKDriverDeaths" "UKgas"
## [93] "USAccDeaths" "USArrests"
## [95] "UScitiesD" "USJudgeRatings"
## [97] "USPersonalExpenditure" "uspop"
## [99] "VADeaths" "volcano"
## [101] "warpbreaks" "women"
## [103] "WorldPhones" "WWWusage"# Para usá-los faça
data("dado")
help("dado")Funções de auto ajuda
# Procura todas as funções com "help"
apropos("help")## [1] "help" "help.request" "help.search" "help.start"# Ajudas genéricas
help.start()
help.search("survival")
# Ajudas expecíficas
help(mean)
example("mean")
# Procura no site do R
RSiteSearch("t.test")Informações da sessão
## R version 3.3.1 (2016-06-21)
## Platform: x86_64-pc-linux-gnu (64-bit)
## Running under: Ubuntu precise (12.04.5 LTS)
##
## locale:
## [1] LC_CTYPE=en_US.UTF-8 LC_NUMERIC=C
## [3] LC_TIME=en_US.UTF-8 LC_COLLATE=en_US.UTF-8
## [5] LC_MONETARY=en_US.UTF-8 LC_MESSAGES=en_US.UTF-8
## [7] LC_PAPER=en_US.UTF-8 LC_NAME=C
## [9] LC_ADDRESS=C LC_TELEPHONE=C
## [11] LC_MEASUREMENT=en_US.UTF-8 LC_IDENTIFICATION=C
##
## attached base packages:
## [1] grid stats graphics grDevices utils datasets base
##
## other attached packages:
## [1] gridExtra_2.2.1 ggplot2_2.2.0 knitr_1.15.1
##
## loaded via a namespace (and not attached):
## [1] Rcpp_0.12.8 revealjs_0.7 assertthat_0.1 digest_0.6.10
## [5] rprojroot_1.1 plyr_1.8.4 gtable_0.2.0 backports_1.0.4
## [9] magrittr_1.5 evaluate_0.10 scales_0.4.1 highr_0.6
## [13] stringi_1.1.2 lazyeval_0.2.0 rmarkdown_1.2 labeling_0.3
## [17] tools_3.3.1 stringr_1.1.0 munsell_0.4.3 yaml_2.1.14
## [21] colorspace_1.3-1 htmltools_0.3.5 tibble_1.2 methods_3.3.1